近日,理学院应用物理系软物质与生物功能材料研究团队在国际TOP学术期刊《International Journal of Biological Macromolecules》 (IF=8.2,中科院一区TOP) 期刊上发表了题为"Geometric deep learning for the prediction of magnesium-binding sites in RNA structures"的研究论文。浙江科技大学为第一单位,应用物理系2021级硕士研究生王康为第一作者,浙江科技大学孙婷婷教授、江苏理工学院许晓军教授为论文通讯作者。

图1 论文发表页面
该研究基于深度学习和分子指纹技术(图2),从RNA-Mg2+三维结合口袋获取点云表面数据,以测地线半径为12Ả提取表面补丁,分别提取形状索引(SI)、距离相关曲率(DDC)、Mg2+与顶点距离(DMV)、Mg2+与顶点角度(AMV)和原子电荷(AC)等四个几何特征和一个化学特征制作分子表面指纹,送入残差网络进行学习和预测,结果如图3所示。
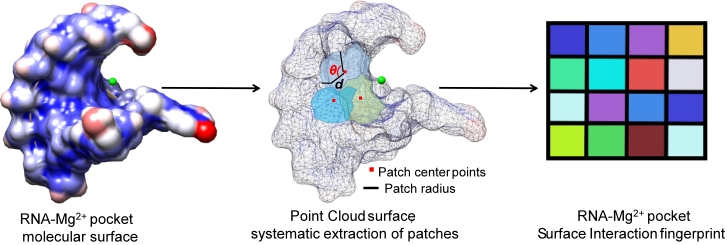
图2 左:RNA-Mg2+结合口袋;中:RNA-Mg2+口袋点云数据表面;
右:分子表面相互作用指纹
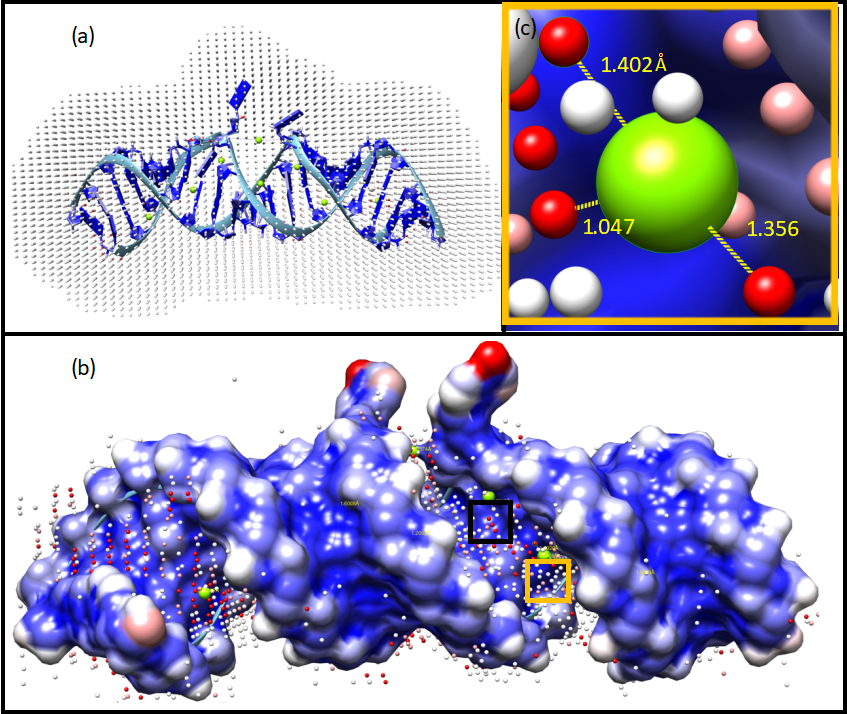

该模型对独立测试集462D的8个实验测定的Mg2+在可信度为TOP-1时,预测精确度为87.5%,在可信度为TOP-3时,精确度为100%。相较于最新的MgNet的TOP-12、Metal3DRNA的TOP-20深度算法,本研究的学习算法有较大提升,而且在捕捉Mg2+与RNA之间的物理相互作用方面特别有效。此项工作得到了国家自然科学基金、浙江省自然科学基金的资助。(应用物理系 孙婷婷 学科办 陈璟)
文章链接:https://doi.org/10.1016/j.ijbiomac.2024.130150